x-meansでクラスタリング【python】
CPPXのXです。
pyclusteringというライブラリを使ってx-meansをやってみたいと思います。 x-meansは、k-meansのkを自動推定しつつクラスタリングしてくれる手法です。
x-meansのアルゴリズムの詳細については、別の方が詳しく説明しています。
環境
- python 3.5.3
- pyclustering 0.8.1
pyclusteringはpipでサクッと入ります。
今回使うもの達
import pyclustering from pyclustering.cluster import xmeans import numpy as np import pylab
2次元データ
とりあえずデータを作ります。
データの持ち方は、(データ数, xy) です。
これをクラスタリングします。
data = np.concatenate((
np.random.uniform(-10, 0, (100, 2)),
np.random.uniform(-30, 0, (100, 2)) + [0, 50],
np.random.uniform(-30, -20, (100, 2)),
np.random.uniform(-60, -30, (100, 2)) + [-50, 30],
np.random.uniform(-50, -40, (100, 2)),
np.random.uniform(-60, -50, (100, 2)) + [50, 0],
np.random.uniform(-70, -60, (100, 2)),
np.random.uniform(-80, -70, (100, 2)) + [-30, 10],
np.random.uniform(-90, -80, (100, 2)),
np.random.uniform(-100, -80, (100, 2)) + [70, 0],
), axis=0)
以下のような適当に作ったデータです。
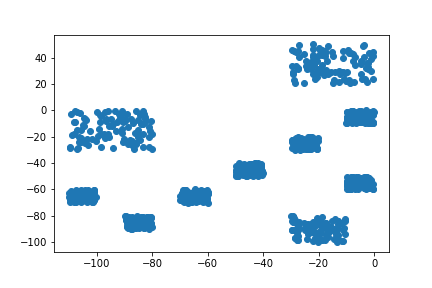
では、クラスタリングします。
初期値は、k-means++による初期値決定を行います。
init_center = pyclustering.cluster.xmeans.kmeans_plusplus_initializer(data, 2).initialize() # 初期値決定 今回は、初期クラスタ2です xm = pyclustering.cluster.xmeans.xmeans(data, init_center, ccore=False) xm.process() # クラスタリングします
ccore=Trueにすると、処理にc/c++が使われて早くなるみたいです。
可視化するには、pyclusteringで用意されてる関数を使うと楽です。
clusters = xm.get_clusters() pyclustering.utils.draw_clusters(data, clusters)
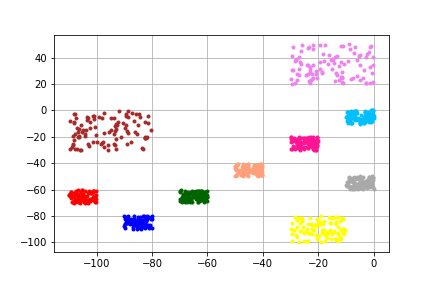
描画にはmatplotlibが使われています。
showさせたくない場合は、display_result=Falseにするといいです。
3次元データ
データを3次元にすると、あとは勝手にやってくれます。
data = np.concatenate((
np.random.uniform(-10, 0, (100, 3)),
np.random.uniform(-20, -10, (100, 3)) + [0, 50, 0],
np.random.uniform(-30, -20, (100, 3)),
np.random.uniform(-40, -30, (100, 3)) + [-50, 20, 30],
np.random.uniform(-50, -40, (100, 3)),
np.random.uniform(-60, -50, (100, 3)) + [50, 0, 100],
np.random.uniform(-70, -60, (100, 3)),
np.random.uniform(-80, -70, (100, 3)) + [-30, 10, -30],
np.random.uniform(-90, -80, (100, 3)) + [0, 0, 50],
np.random.uniform(-100, -90, (100, 3)) + [70, 0, -30],
), axis=0)
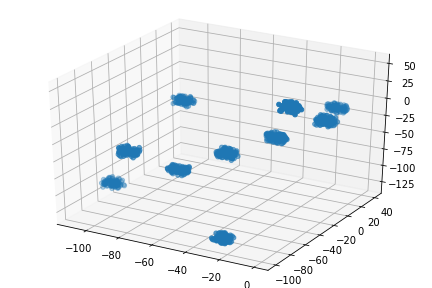
これにx-meansかけると、
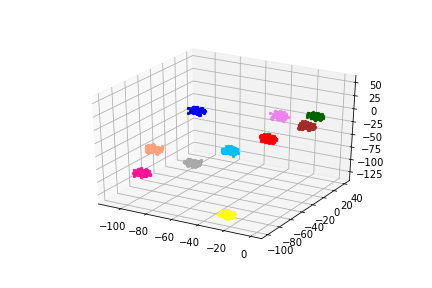
良さげです。
クラスタリング部分のソースコードは2次元の時に使ったものそのままで大丈夫です。
描画は1~3次元までしか出来ませんが、クラスタリング自体は何次元でも出来ると思います。
irisデータセットの一部にやってみる
可視化する関係上、使うデータを以下の3つに絞ります
- sepal length (cm)
- sepal width (cm)
- petal length (cm)
from sklearn import datasets iris = datasets.load_iris() iris.data.shape, iris.target.shape using = (0, 1, 2) data = iris.data[..., using]
正解ラベル付きで表示しています。
このデータにx-meansをあててみます。


ほう
今回自分が使ったコードまとめ
おわりです
最後に、今回使ったコードをまとめて貼っておきます。
グラフ表示部分も含めておきました。
2次元
#%% imports import pyclustering from pyclustering.cluster import xmeans import numpy as np import pylab #%% create data 2d data = np.concatenate(( np.random.uniform(-10, 0, (100, 2)), np.random.uniform(-30, 0, (100, 2)) + [0, 50], np.random.uniform(-30, -20, (100, 2)), np.random.uniform(-60, -30, (100, 2)) + [-50, 30], np.random.uniform(-50, -40, (100, 2)), np.random.uniform(-60, -50, (100, 2)) + [50, 0], np.random.uniform(-70, -60, (100, 2)), np.random.uniform(-80, -70, (100, 2)) + [-30, 10], np.random.uniform(-90, -80, (100, 2)), np.random.uniform(-100, -80, (100, 2)) + [70, 0], ), axis=0) pylab.scatter(data[..., 0], data[..., 1]) pylab.savefig("./data.png") pylab.show() #%% clustering init_center = pyclustering.cluster.xmeans.kmeans_plusplus_initializer(data, 2).initialize() xm = pyclustering.cluster.xmeans.xmeans(data, init_center, ccore=False) xm.process() clusters = xm.get_clusters() pyclustering.utils.draw_clusters(data, clusters, display_result=False) pylab.savefig("./data2.png") pylab.show()
3次元
#%% imports import pyclustering from pyclustering.cluster import xmeans import numpy as np import pylab from mpl_toolkits.mplot3d.axes3d import Axes3D import matplotlib import matplotlib.pyplot as plt #%% create data 3d data = np.concatenate(( np.random.uniform(-10, 0, (100, 3)), np.random.uniform(-20, -10, (100, 3)) + [0, 50, 0], np.random.uniform(-30, -20, (100, 3)), np.random.uniform(-40, -30, (100, 3)) + [-50, 20, 30], np.random.uniform(-50, -40, (100, 3)), np.random.uniform(-60, -50, (100, 3)) + [50, 0, 100], np.random.uniform(-70, -60, (100, 3)), np.random.uniform(-80, -70, (100, 3)) + [-30, 10, -30], np.random.uniform(-90, -80, (100, 3)) + [0, 0, 50], np.random.uniform(-100, -90, (100, 3)) + [70, 0, -30], ), axis=0) fig = plt.figure() ax = Axes3D(fig) ax.scatter3D(data[..., 0], data[..., 1], data[..., 2]) pylab.savefig("./data.png") plt.show() #%% clustering init_center = pyclustering.cluster.xmeans.kmeans_plusplus_initializer(data, 2).initialize() xm = pyclustering.cluster.xmeans.xmeans(data, init_center, ccore=False) xm.process() clusters = xm.get_clusters() pyclustering.utils.draw_clusters(data, clusters, display_result=False) pylab.savefig("./data2.png") pylab.show()
iris
#%% imports import pyclustering from pyclustering.cluster import xmeans import numpy as np import pylab from mpl_toolkits.mplot3d.axes3d import Axes3D #%% load data from sklearn import datasets iris = datasets.load_iris() iris.data.shape, iris.target.shape #%% plot iris data using = (0, 1, 2) iris.feature_names data = iris.data[..., using] target = np.concatenate([iris.target[np.where(x == iris.target)] for x in using]) fig = pylab.figure() ax = Axes3D(fig) ax.scatter3D(data[..., 0], data[..., 1], data[..., 2], c=iris.target) pylab.savefig("./data.png") pylab.show() #%% clustering 3 data init_center = pyclustering.cluster.xmeans.kmeans_plusplus_initializer(data, 2).initialize() xm = pyclustering.cluster.xmeans.xmeans(data, init_center, ccore=False) xm.process() clusters = xm.get_clusters() print(len(clusters)) pyclustering.utils.draw_clusters(data, clusters, display_result=False) pylab.savefig("./data2.png") pylab.show()